Introducción:
En 2017 se reportó la emergencia del clon de alto riesgo de K. pneumoniae (Kp) ST307 en Argentina. Kp-ST307 rápidamente se estableció en entornos hospitalarios, y en 2019 constituyó el 30% del total de aislamientos de Kp recuperados en 2 hospitales de AMBA.
Objetivos:
Describir las características genómicas de 9 aislamientos de Kp ST307 productores de carbapenemasas recuperados de 3 hospitales de AMBA y establecer su relación filogenética con otros reportados a nivel mundial.
Materiales y métodos:
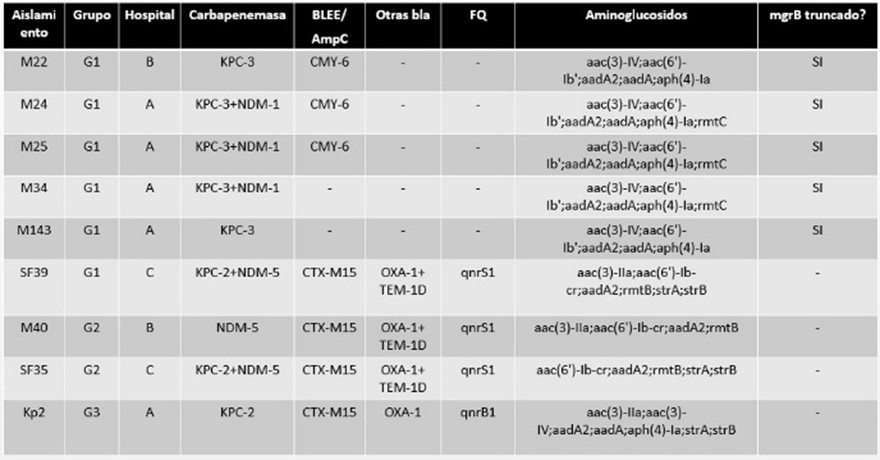
Se analizaron 7 aislamientos (SF35, SF39, M24, M25, M34, M40 y M143) recuperados en 3 hospitales durante 2018-21, y 2 aislamientos originales de Kp-ST307 recuperados en 2017 (M22 y Kp2). La sensibilidad a los antimicrobianos se determinó según los criterios del CLSI. Se realizó secuenciación de genoma completo (WGS) sobre el ADN total utilizando Illumina MiSeq. Las reads se ensamblaron utilizando SPAdes y se anotaron utilizando PROKKA. Se utilizó PathogenWatch (Kleborate, PlasmidFinder, ResFinder y Phylocanvas) para el análisis bioinformático. Para el análisis filogenético se seleccionaron 250 genomas de Kp-ST307disponibles en NCBI y los 9 aislamientos mencionados anteriormente. El filoanálisis se construyó utilizando Roary, SNPsites, IQtree, iTOL, y se visualizó utilizando Microreact.
Resultados:
El resultado del análisis bioinformático se muestra en la tabla. En las Kp-ST307 portadoras de blaNDM-5 y blaNDM-1 se detectaron los genes rtmB y rtmC de resistencia a aminoglucósidos, respectivamente. En todas las Kp-ST307 se predijo wzi173 relacionado con el tipo capsular KL102 y el tipo O2. Excepto en SF39 que albergó ybt9, no se detectaron genes de virulencia accesorios,
Discusión / Conclusiones:
Las Kp-ST307 de Argentina mostraron pequeñas diferencias en cuanto a su resistoma y su relación filogenética dentro del clado C1.