Introducción
El complejo clonal 258 (CC258) de Klebsiella pneumoniae (Kpn), con sus principales secuenciotipos ST258 y ST11, ha sido el principal responsable de la diseminación global de KPC. Los métodos tradicionales para identificar los clones hiperepidémicos como PFGE, MLST, WGS, son laboriosos, costosos y poco accesibles para los paises de bajos y medianos ingresos (LMIC). La proteína PilV podría estar involucrada en la biogénesis de fimbrias, patogenicidad y virulencia. En un estudio previo, se demostró que una PCR dirigida al gen de la proteína PilV (pilV-l) tuvo una sensibilidad y especificidad de 100% para identificar Kpn ST258 (Gómez S., Infect Genet Evol. 2016). La capacidad de adaptación y evolución de Kpn resalta la importancia de evaluar los métodos moleculares para garantizar su efectividad a lo largo del tiempo.
Objetivo
Evaluar mediante análisis in silico la capacidad de la PCR de pilV-l para la identificación del clon de Kpn ST258 en aislamientos contemporáneos de Kpn y determinar su efectividad como método de tipificación.
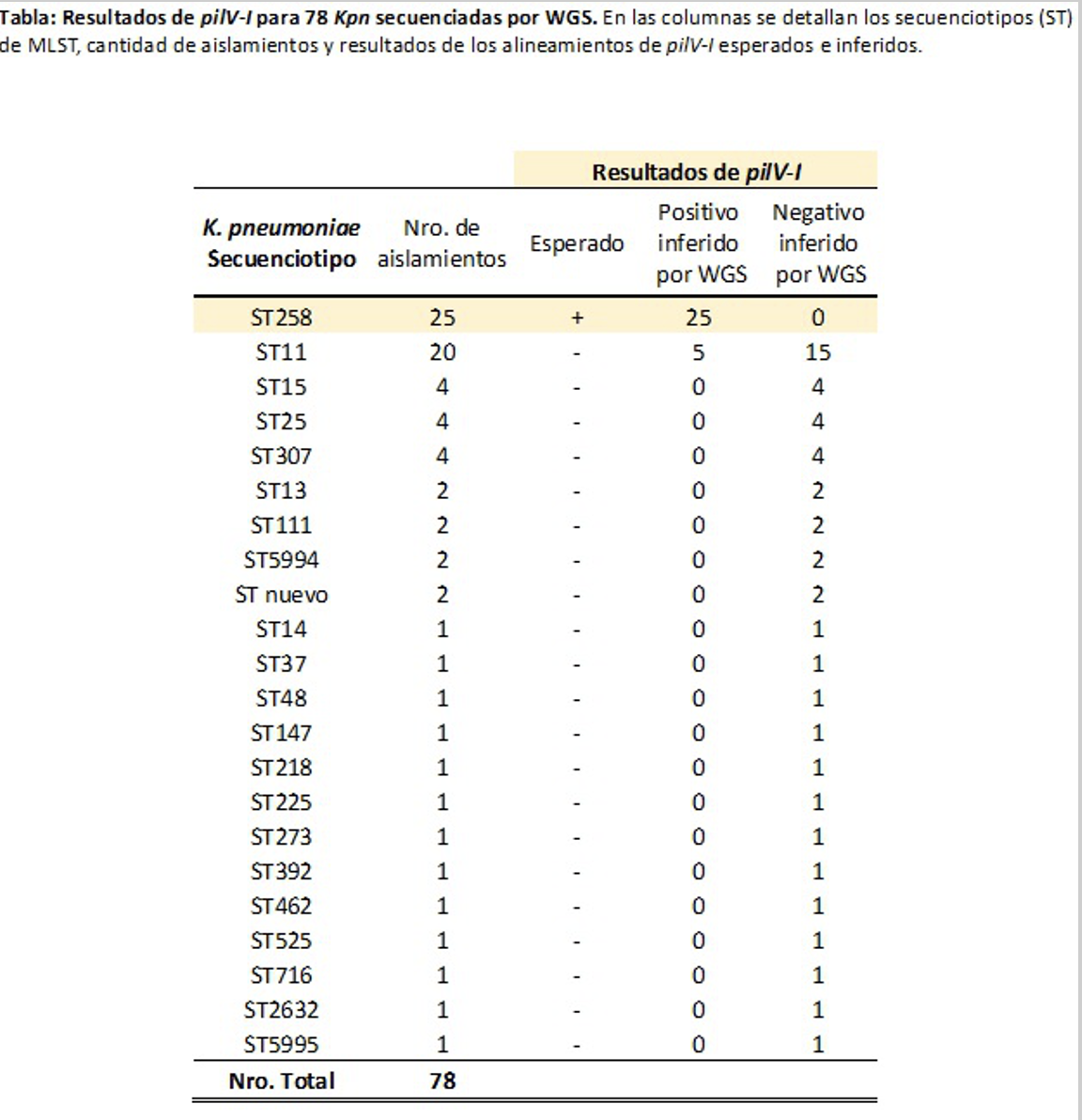
Materiales y Métodos: Se secuenciaron por Illumina una selección de 78 Kpn, provenientes de un Corte Nacional de Prevalencia de Enterobacterales portadoras de Carbapenemasas (RECAPTAR I) de infecciones, realizado en noviembre de 2021, que incluyó 183 hospitales y 23 jurisdicciones. La selección incluyó 25 STs: ST258 (n=25) y ST11 (n=20) del CC258 y 23 STs no-CC258 (n=33) (Tabla). Las carbapenemasas se analizaron con AMRFinderPlus, la PCR in silico de pilV-l se infirió en base a los alineamientos usando el algoritmo NCBI-BLAST y el MLST mediante https://pubmlst.org/.
Resultados
Las 25 Kpn del ST258, portadores de NDM-5 (16), KPC-2 (7), NDM-1 (1) y OXA-163 (1), alinearon con el amplicón esperado de PCR para pilV-l. De las 20 Kpn ST11, 5 dieron falso positivo para pilV-l, posiblemente debido a que el ST11 es una variante de locus único del ST258 y forma parte de este CC. Entre las 33 Kpn del grupo no-CC258, 31 no alinearon con pilV-l y sólo 2 lo hicieron de manera incompleta, por lo que se esperaría que no amplifique en un ensayo de PCR.
Conclusiones
Este estudio confirma que el ensayo de PCR de pilV-l presenta una SE de 100% y ES de 90,6% para la identificación del ST258 en aislamientos contemporáneos. Este ensayo continúa siendo un método práctico y de bajo costo para la implementación y seguimiento de este clon hiperepidémico en LMICs.

