Introducción:
La hepatotoxicidad inducida por fármacos antituberculosis (HIFA) es una reacción adversa frecuente, grave y potencialmente fatal del tratamiento de la tuberculosis (TB) que conlleva a que aproximadamente un 11% de los pacientes discontinúen el tratamiento. Ciertas variaciones genéticas en las enzimas que metabolizan Pirazinamida (PZA), fármaco anti-TB, podrían producir enzimas defectuosas con la consecuente acumulación tóxica del fármaco y sus metabolitos.
Objetivo:
Evaluar la asociación de factores ambientales y variantes genéticas en enzimas que metabolizan PZA con HIFA. Investigar interacciones gen-gen y gen-ambiente y su asociación con HIFA, en una población de pacientes con TB hospitalizados de la Ciudad de Buenos Aires (Argentina).
Materiales y Métodos:
Estudio de casos y controles (2017-2019). Se analizaron variables clínicas y demográficas de 340 pacientes con TB (95 con HIFA). Se analizó el genotipo de las variaciones polimórficas (SNP) (rs1054889, rs6752058, rs6710015 y rs1042039) de la enzima Xantin oxidasa (XO) por secuenciación genómica. Se compararon las posibles variables predictoras entre pacientes con (caso) y sin (control) HIFA por regresión logística binaria. Se estudiaron las interacciones genéticas y ambientales en asociación con HIFA por el método de reducción de la dimensionalidad multifactorial.
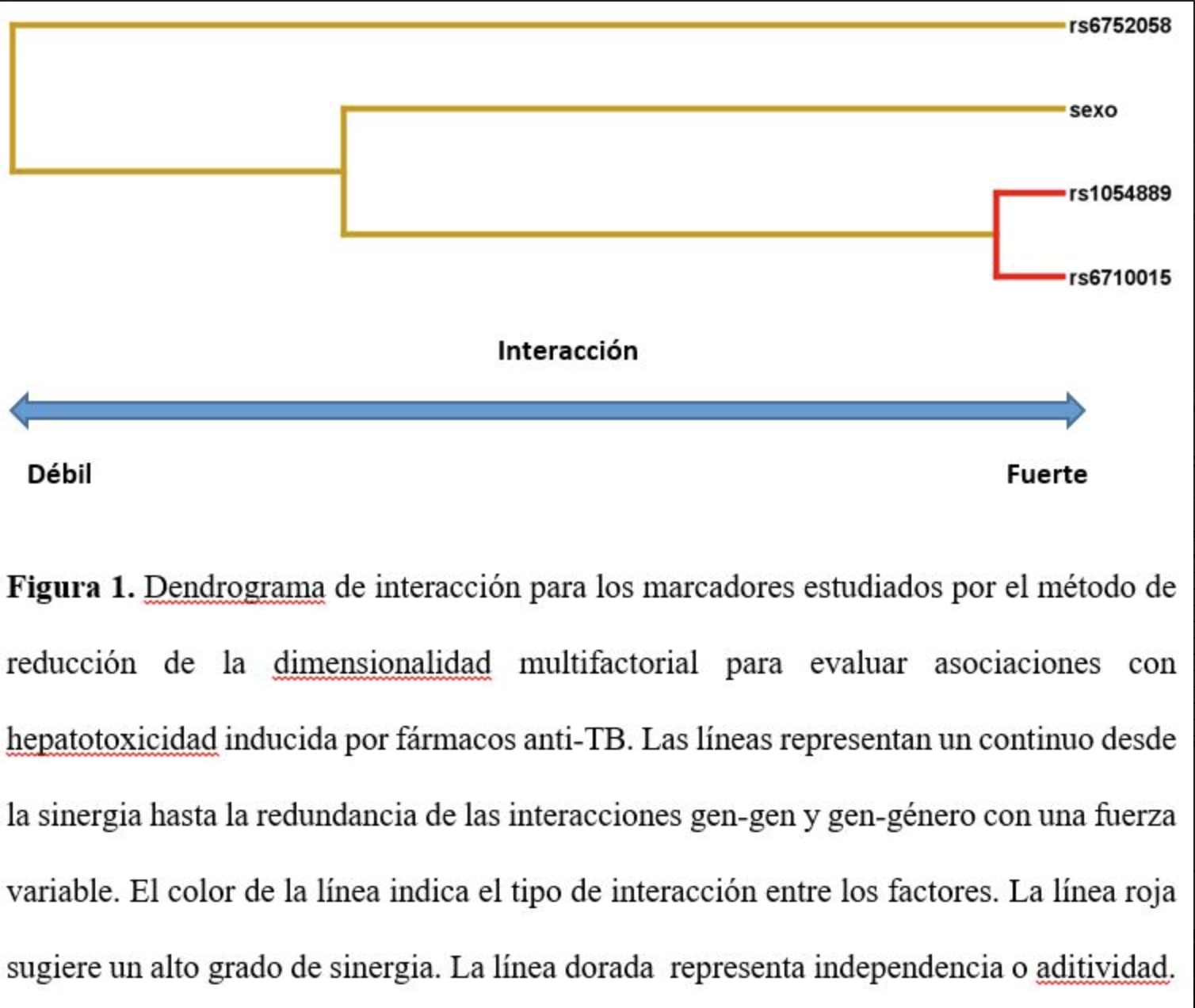
Resultados:
No se hallaron diferencias significativas (p>0.05) para ningún SNP entre los pacientes con y sin HIFA. Tampoco se encontró asociación entre los genotipos analizados con HIFA. Al analizar las interacciones génicas, se identificó por primera vez una interacción sinérgica (epistasis) entre rs1054889 y rs6710015 con mayor riesgo de HIFA (figura 1). Sin embargo, no se halló ningún modelo de predicción superador de los estudiados anteriormente con otras enzimas.
Conclusiones:
Si bien se halló una interacción génica, por primera vez descripta, no hemos encontrado ninguna variable predictora que arroje un modelo de predicción de HIFA superador al modelo hallado previamente que contempla las variables génicas de NAT2 y CYP2E1 y a la variable ambiental, sexo. Hasta el momento, este es el primer trabajo en estudiar polimorfismos de la enzima XO y el desarrollo de HIFA.

