Introducción
El FilmArray™ (FA) es una herramienta de diagnóstico microbiológico basada en PCR multiplex con paneles sindrómicos (neumonía, BCID2, meningitis/encefalitis, osteoarticular y gastrointestinal) para la detección de los patógenos más relevantes al cabo de una hora. Permite adecuar precozmente los tratamientos antimicrobianos empíricos (TE), optimizando su uso. El objetivo de este estudio fue describir la correlación con los métodos de cultivo y su rol en la adecuación de los tratamientos antibióticos.
Materiales y métodos
estudio observacional, descriptivo, de cohorte retrospectiva que incluyó a pacientes mayores de 18 años de edad hospitalizados con muestras analizadas por FA y cultivo entre el 01/09/2023 y el 31/05/2024 en un hospital general de agudos de segundo nivel de la ciudad de Buenos Aires.
Resultados
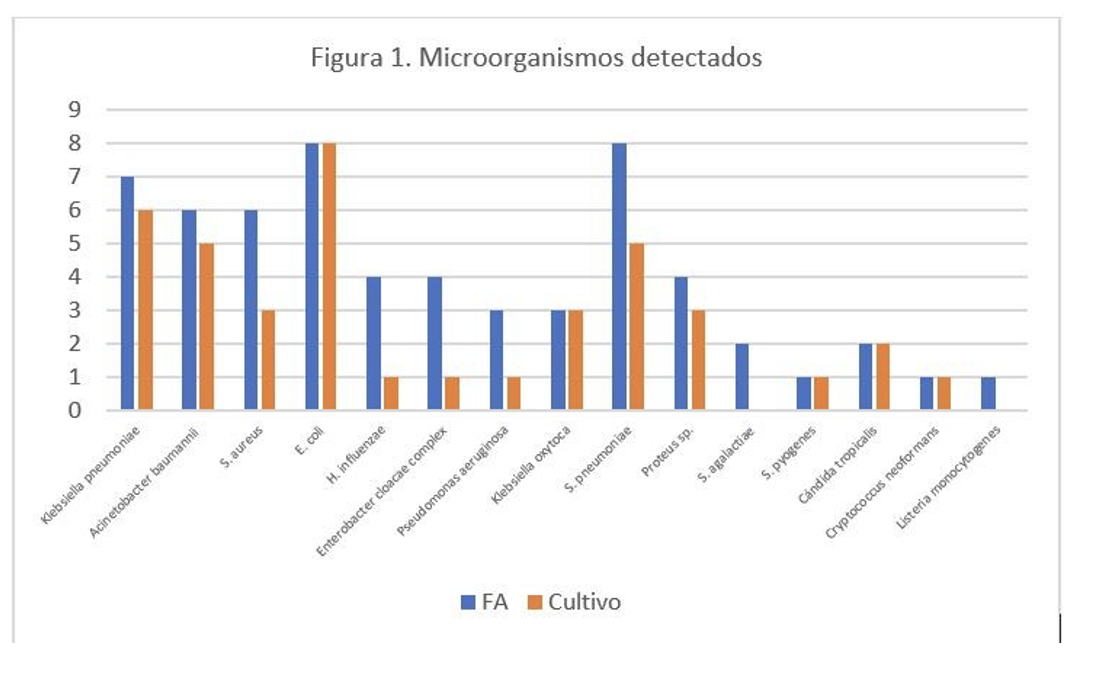
se incluyeron 49 pacientes y 62 muestras (11 pacientes aportaron dos o más muestras). El 57,1% (n= 28) eran varones. La media de edad fue de 56,41 años (DE +- 19,6). El 65% (n= 32) tenía comorbilidades. El 60% de los pacientes (n= 29) se encontraba internado en guardia o sala general. Las infecciones más frecuentes fueron la NAC (n= 12), la NIH (n=7), bacteriemia primaria (n= 7) y secundaria (n= 10) y la meningitis (n= 2). Las muestras analizadas fueron sangre (n= 28), aspirado traqueal (n= 16), LCR (n= 11), BAL (n= 3) y esputo (n= 3). Los microorganismos detectados tanto en Film Array como en cultivo se muestran en la figura 1. En el 75% (n= 46) de los casos hubo concordancia de detección entre FA y cultivo. La media de tiempo hasta obtener el informe del cultivo fue de 2,85 días (DE +- 1,55 días).El FA detectó genes de resistencia en el 28% (n=16) de los casos, siendo los más frecuentes mec A/C (n=5), KPC (n= 3), NDM (n= 3) y CTX-M (n=5), con concordancia con los mecanismos de resistencia detectados por cultivos. El resultado del FA permitió modificar el TE en el 60% (n= 37) de los casos, siendo el desescalamiento el cambio más frecuente (57%, n= 21). La letalidad fue de 37% (n= 18) sin diferencia significativa entre el grupo al cuál se le modificó el TE ante el resultado del FA con respecto al que no (p= 0.47).
Conclusiones
la implementación del FA en nuestro centro, nos permitió mejorar los tiempos de diagnóstico y de adecuación de los tratamientos antibióticos con una alta concordancia con los resultados de los cultivos y una rápida detección de los genes de resistencia antibiótica.